一、输入序列,选择数据库,设置参数后提交任务。
1. 在“服务首页”页面,如下所示的框内输入要查询的 DNA 序列。
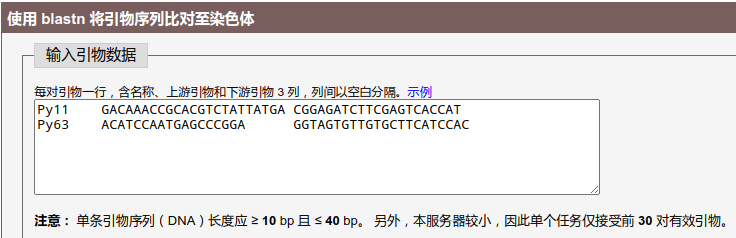
每行粘贴 1 对引物的信息,包含以空白字符(如水平制表符 Tab)分隔的 3 列,第 1 列为引物编号,第 2 列为上游引物序列,第 3 列为下游引物序列。单条引物序列长度可修改,当前服务器设置为 ≥ 10 bp 且 ≤ 40 bp。
2. 选择要 blast 的数据库。
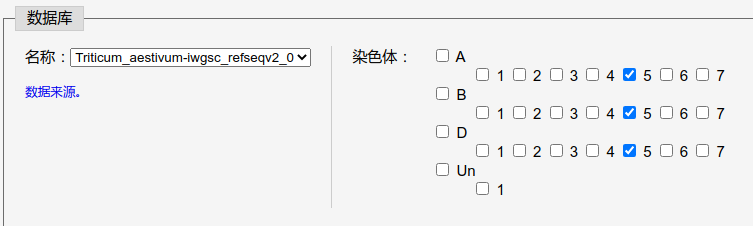
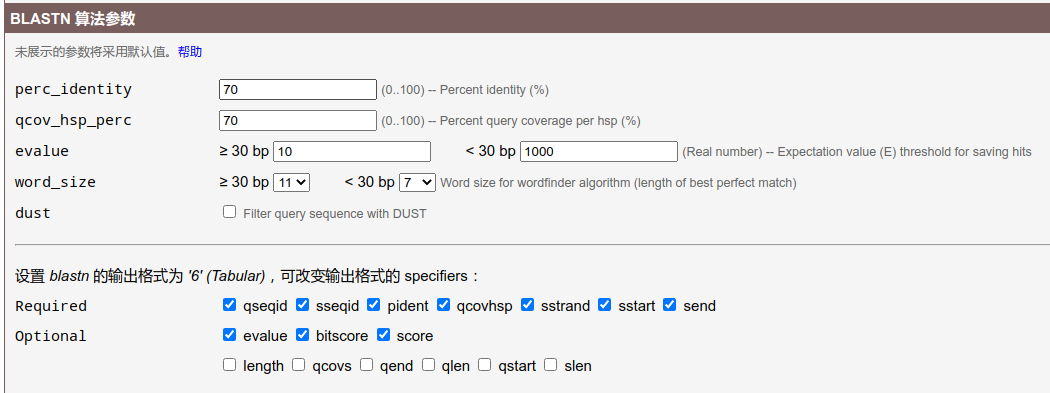
图中物种选择了小麦 IWGSC RefSeq v2.0 参考基因组的 5 同源群染色体,那么任务提交后会将序列和这 3 条染色体序列分别进行比对。至于其他参数(“BLASTN 算法参数”),可以根据需要设定,这里直接用默认的。其中,perc_identity 和 qcov_hsp_perc 越大,对匹配序列和查询序列的相似度要求越高。evalue(要大于 0)越小,对相似度的要求越高。这些参数都是 blastn 程序的,点击图中链接会访问 NCBI 的相关用户手册。
3. 提交任务。
设定妥当,点击 BLASTN 按钮,提交任务。
点击后将跳转至“任务状态”页面。
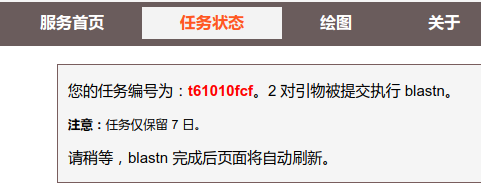
页面会自动刷新,直至任务完成。图中给出了一个“任务编号”,也就是 t61010fcf,你可以记录下它,稍后访问 任务状态,根据这个 Id 来查询并操作。
某些物种(例如小麦)基因组很大,若服务器配置较低,blastn 就可能得跑比较久。之所以限定最多允许一次检索 30 条序列,也是怕服务器伤不起。
二、根据 blast 结果进行分析。
1. Blastn 结束后任务状态页面 + 选项卡 + 查看 Blast 结果。
Blastn 结束后,页面会如下图所示。
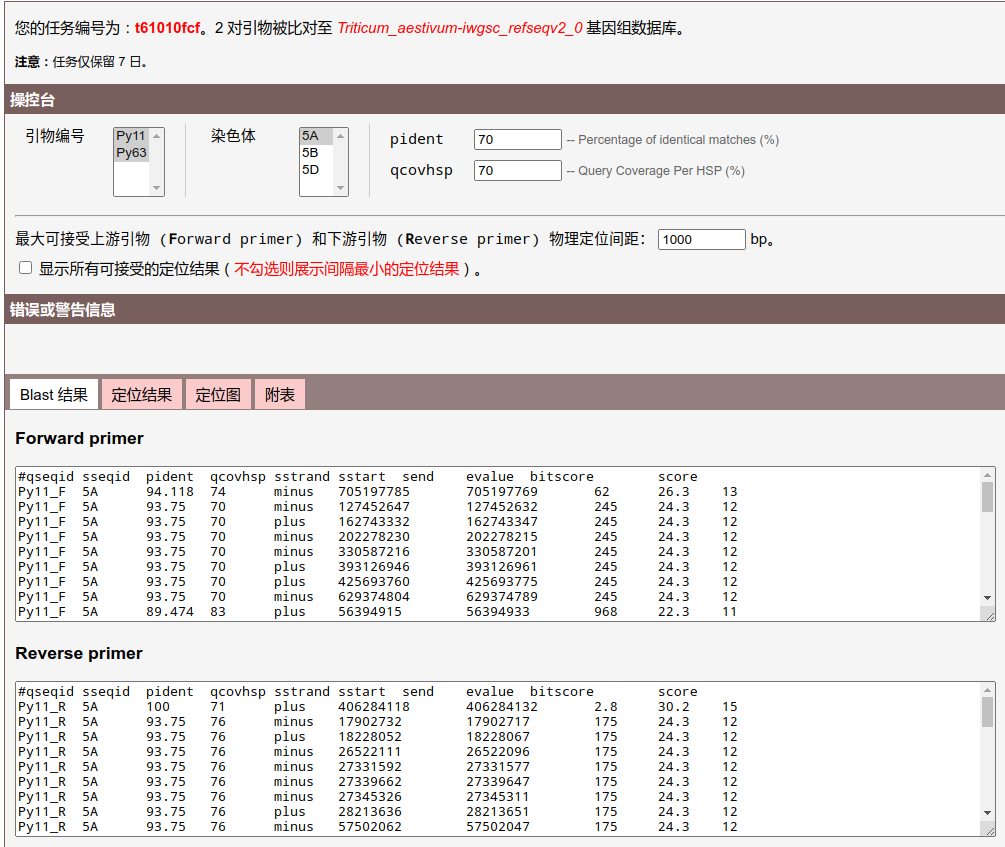
上图的顶部给出了任务编号、比对的引物对数和参考基因组,以及当前服务器对临时任务的保留时长:7 日。
上图的中部为“操控台”,修改参数后“错误和警告信息”和图底部结果选项卡可以即时响应。“操控台”中,“引物编号”可多选,“染色体”只能单选,pident、qcovhsp 和“最大可接受物理定位间距”请勿输入负数。
“错误和警告信息”模块将显示错误(字体为红色)和警告(字体为黑色)。
上图的底部有 4 个选项卡,分别为“Blast 结果”“定位结果”“定位图”和“附表”。鼠标左键单击相应选项卡,可看到相应的结果、图表。
页面刚刷新出来时,会展示“Blast 结果”,其中“Forward primer”下面的文本框内展示上游引物的 blast 结果,“Reverse primer”下的文本框则展示下游引物的 blast 结果,若有需要,可以选中后复制保存至文档。
2. 查看定位结果。
5A
5B

5D


5D

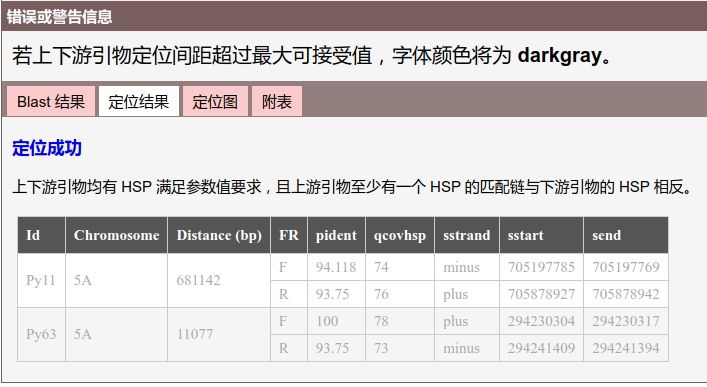
如上面 3 张图所示,点击“定位结果”选项卡,再分别点击“染色体”列表中的 3 条染色体,观察可知 2 对引物均是在 5B 上有较好的定位,可能扩增出长度合适(1,000 bp 及以下)的产物。5A 和 5D 则无法很好的定位,可能扩增不出长度合适的产物,相应的,根据“错误和警告信息”指示,此时,虽然展示了“定位成功”的结果,但是上下游引物定位距离过远,因此显示为黑灰色,即虽有定位,但可能不合适。
若存在引物对只有一条序列可以定位,或两条序列均无法定位,将展示“定位失败”的引物编号表。

单击“定位成功”,可以对表格内容进行全选,方便复制。

若勾选“显示所有可接受的定位结果(不勾选则展示间隔最小的定位结果)”,则“错误和警告信息”显示“未发现可接受的定位结果”。此复选框主要针对随机引物,其可能在同一染色体上有多处扩增,勾选则全部展示,否则只展示间距最小的定位结果。
5D - 勾选“显示所有可接受的定位结果”

若“定位成功”,且上下游引物定位间距在限定范围内,则可以单击相应“Id”(下图红色箭头指示),查看定位区间的序列。

3. 查看定位图。
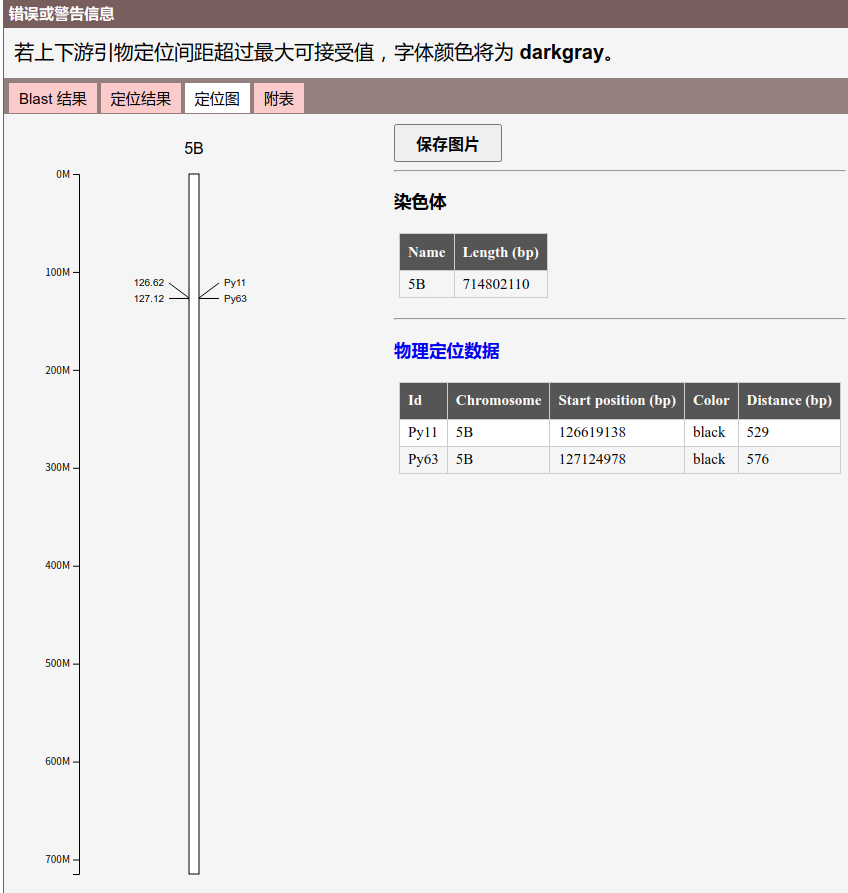

如上图所示,点击“定位图”选项卡,可绘制出 2 对引物的定位图,可对图片进行保存,鼠标悬浮在定位图中的分子标记编号、物理位置和枝杈上时,将展示出其具体的定位坐标和上下游引物定位间距。同时,在图片右侧给出了染色体的长度和定位表,点击“物理定位数据”可以全选相应表格内容,方便复制。
可将“定位图”展示的染色体长度和物理定位数据保存汇总,而后在“绘图”页面进行进一步作图。
4. 查看附表。

如上图,点击“附表”选项卡,可查看任务的相关参数。
本服务提供了一个专门的绘图页面,方便:(1)对由本服务产生的多组数据汇总画图,(2)对从其他地方获取的数据进行绘图。
如下图所示,该页面包括 3 大块:
- 参数设定选项卡集合。
- 错误和警告信息。
- 绘图结果。
1. “染色体”选项卡。
如上图,用户需要填写 2 块内容:
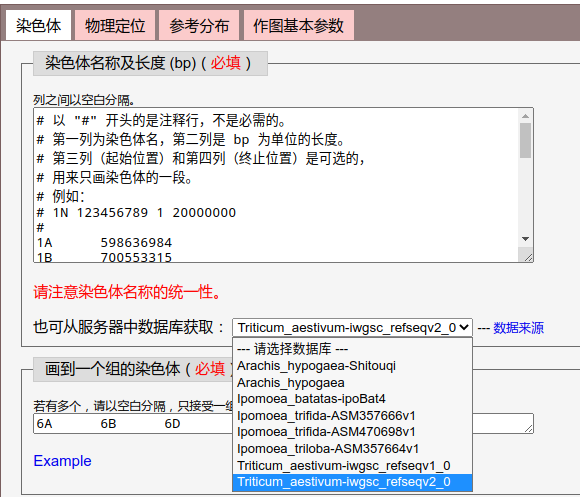
- 染色体名称及长度:文本框中给出了数据格式说明,即“#”号开头的前几行。用户可以填入自己的数据,或者直接点选下拉列表来直接从服务器数据库获取数据。注意,此处文本框中的染色体名称需要和“画到一个组的染色体”“物理定位”“参考分布”中的染色体名称保持一致。
- 画到一个组的染色体:需要填入一个或多个染色体名称,只支持一组染色体,即画图的时候画在一行上。上图为点击“Example”链接使用了示例数据。
2. “物理定位”选项卡。
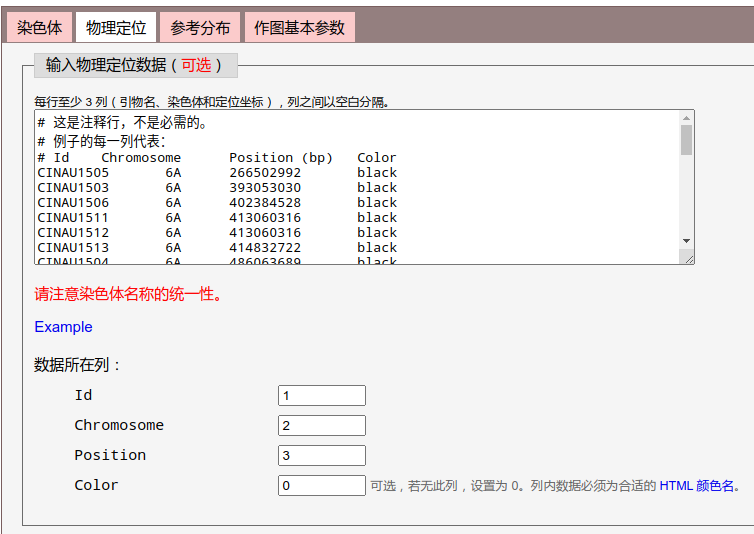
用户需要提供至少 3 列数据:编号(Id)、染色体(Chromosome)和物理定位坐标(Position),并指明对应的列数。上图为点击“Example”链接使用了示例数据。注意,“染色体名称”需要与其他地方填入的数据保持一致。
Color 列为可选,可填入合适的 HTML 颜色名;Color 所在列设为 0 时,将直接使用默认颜色。
3. “参考分布”选项卡。

该选项卡内容为可选,主要是为了方便在图中指示着丝粒大致位置。
用户需要提供 3 列数据:染色体名称、位置坐标和丰度。其中,“位置坐标”列为区段坐标,例如为 1 Mb、500 Kb、100 Kb 等,可以设定其单位;“丰度”列则表示在相应区段内参考序列的丰度,例如着丝粒特异探针序列在 1 Mb 区段的同源拷贝数。
上图使用了“Example - wheat Oliogo-CCS1 (IWGSC RefSeq v2.0)”这一示例数据,即由 MCGB-B2DSC 生成的 Oliogo-CCS1 在 IWGSC RefSeq v2.0 参考基因组中的分布数据。
4. “作图基本参数”选项卡。
可以调整“作图基本参数”,改变绘图结果。其中:
- 整张图“总宽度”如果容不下预备绘制图像,将更新为最小能容下的宽度。
- 整张图“内边距”是为标尺、染色体名称、连接线等预留空间。
- 染色体“末端圆弧半径”调整后可以将染色体端部绘制成圆弧状。
- 如果填入了负数,将在“错误和警告信息”模块提示,并将对应输入框标红。
各项参数设置好后,点击“画图”按钮,进行绘图。

如果绘图成功,将显示“保存图片”按钮,同时,“错误和警告信息”将显示绘图所使用的刻度单位。
5. 绘图结果。

图中染色体上的红色横线指示了着丝粒的大致位置。
可以通过修改参数,调整绘图结果。点击“保存图片”,将得到 SVG 格式图片。